会议回顾
2025年3月27日,应Guangzhou RNA Club邀请,以色列耶路撒冷希伯来大学Orna Amster-Choder教授作了题为“Revisiting the dogma on bacterial transcriptome organization: implications on gene expression”的线上学术报告。Orna Amster-Choder教授的主要研究方向包括:(1)亚细胞组织;(2)相分离;(3)转录-翻译;(4)抗生物膜因子。
会议内容
本次报告围绕细菌 RNA 的定位、转录与翻译的关系、细菌细胞极性相关的 RNA 定位机制、小 RNA 的定位及作用,以及 RNA 伴侣蛋白的相关研究展开,对细菌转录组织的传统理论进行了重新审视,为相关领域研究提供了新视角。
一、细菌 RNA 的特殊时间组织及其生物学意义
1.细菌细胞的传统观点与更新:
传统观点:
传统观点认为细菌中 mRNA 仅编码蛋白质且不发生定位。
新发现:
RNA 定位模式:RNA 分子可在大肠杆菌细胞中定位于特定区域,mRNA 有细胞质、膜、极区三种定位模式,且其定位与编码蛋白定位相关,同一操纵子顺反子的 mRNA 会共同定位 ,且定位独立于翻译。
转录与翻译空间分离:大肠杆菌中转录和翻译机器在空间上分离,少量核糖体和 RNA 聚合酶分布特殊。此外,还涉及大肠杆菌中编码 SRP 插入小膜蛋白的 mRNA、蓝细菌中编码类囊体膜蛋白的 mRNA,均存在独立于翻译的定位现象 。
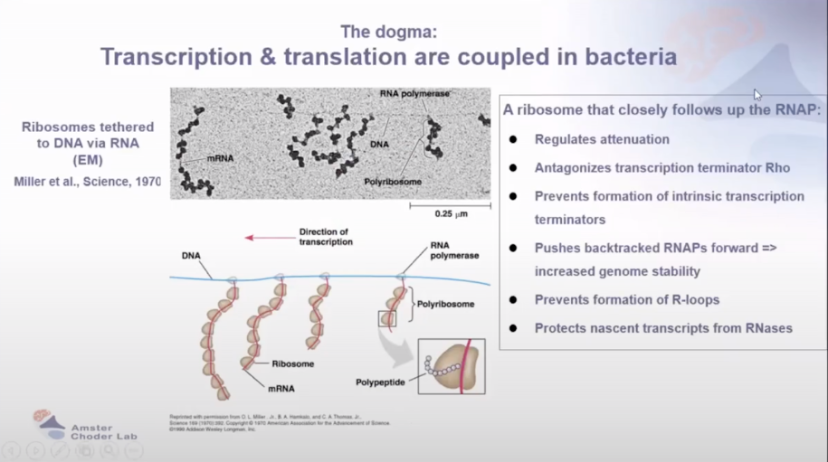
图1 传统观点
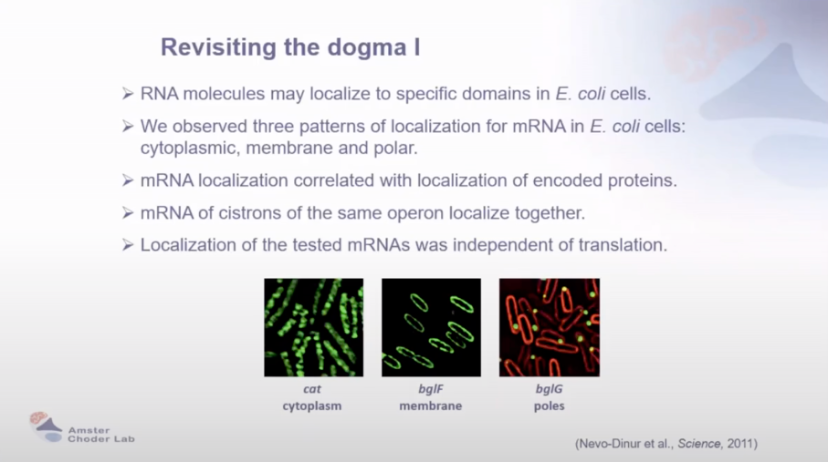
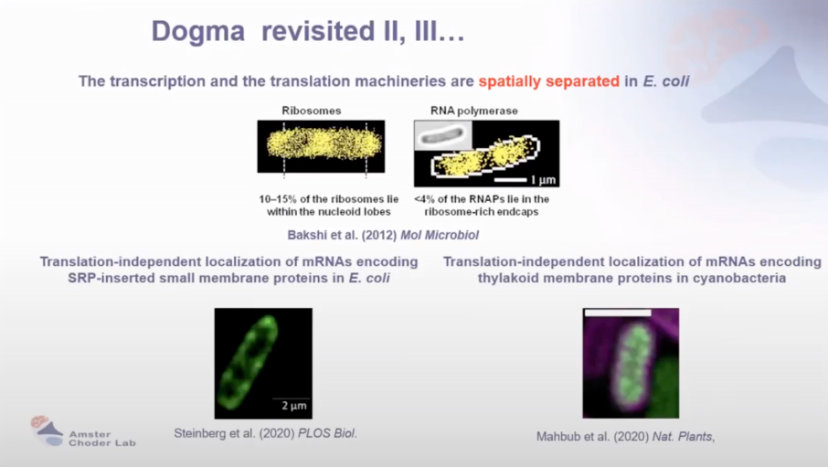
图2 对传统观点的重新审视
2.高通量分析
分级分离与测序:Rloc-Seq将细胞分离为膜、细胞质和极区组分,随后进行 RNA 测序。结果显示,大部分转录组呈现不对称分布,许多 mRNA 在各组分中显著富集。
基因本体分析:分析表明各组分中富集不同功能的基因群。细胞膜组分中富集转运活性、外膜和脂多糖生物合成相关基因;细胞质组分中则存在 DNA 复制和糖酵解相关基因。
翻译独立性:多数 mRNA 的定位不受翻译抑制的影响。例如,用抗生素(如卡斯托霉素)处理或进行移码突变,均未阻止 mRNA 定位于相应区域。
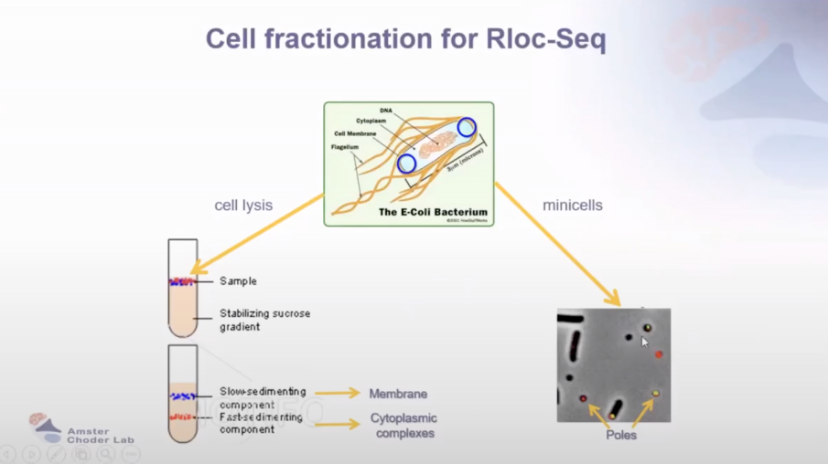
图3 用于 Rloc-Seq 的细胞分离
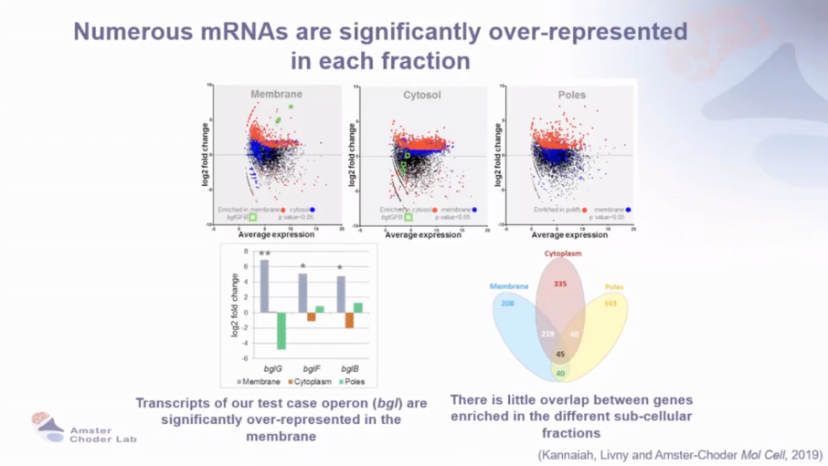
图4 大量 mRNA 在每个级分中显著过度表达
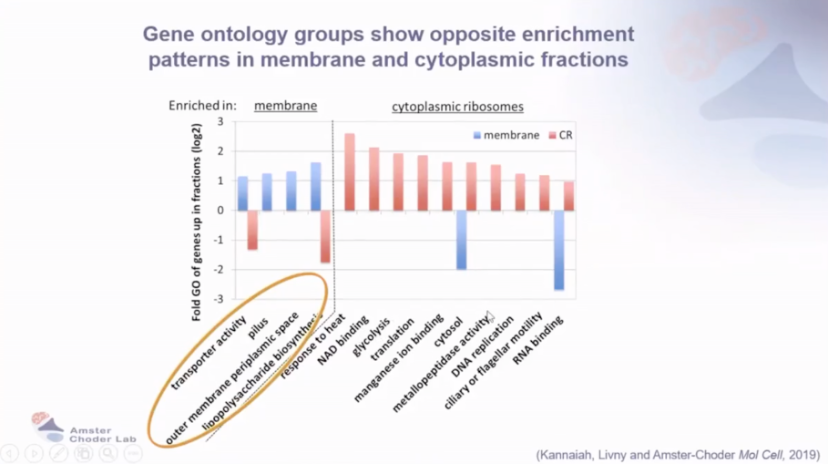
图5 基因本体组在膜和细胞质组分中显示出相反的富集模式
二、RNA 定位在多大程度上取决于翻译?
1.RNA的亚细胞分布与翻译无关性
研究发现,RNA的亚细胞分布(如膜定位)在很大程度上与翻译过程无关。即使抑制翻译起始或引入移码突变,RNA仍能定位到膜上。RNA的膜定位依赖于富含A/U的序列(“zip code”),通过调整这些序列的富集程度可以调控RNA的定位。
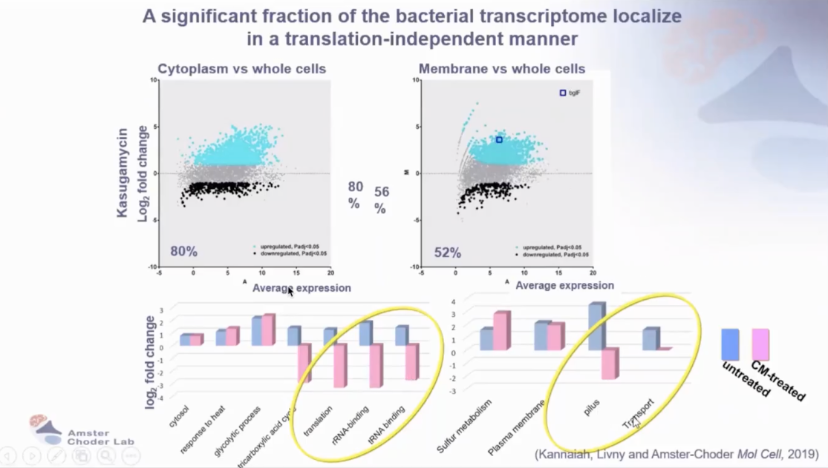
图6 细菌转录组中很大一部分以翻译无关的方式定位
2.极区 mRNA 定位的机制研究
Orna教授及学生采用了两种方法来确定mRNA定位到极点的位置,分别是:采用候选系统方法(基于假设,研究关键蛋白缺失或失活对极区 mRNA 定位的影响)和无偏蛋白质组学方法(通过纯化与 RNA 结合的蛋白,分析相关蛋白)。研究发现,RNA能够特异性地定位到细胞的极区,这一过程依赖于MinD和RNase E的相互作用。RNase E与MinD相互作用,其膜结合域(Segment A)是相互作用的关键区域。
当MinD或RNase E缺失时,极区RNA的定位被破坏,进而影响编码蛋白质的定位。
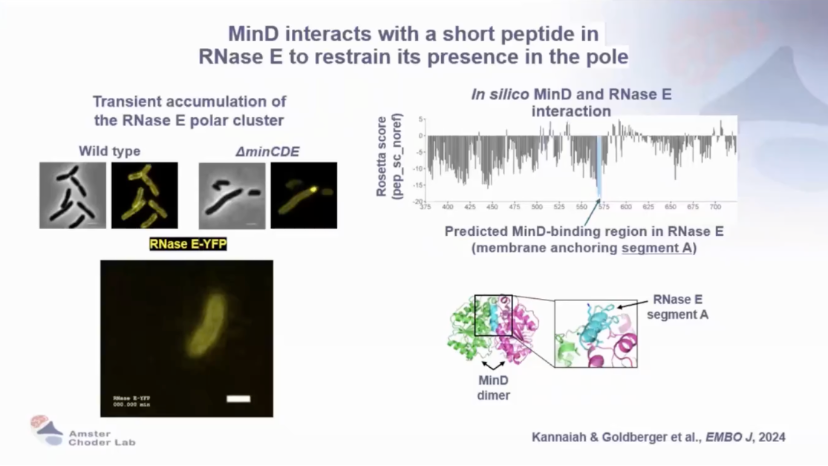
图7 MinD与RNase E中的短肤相互作用,以限制其在极点的存在。
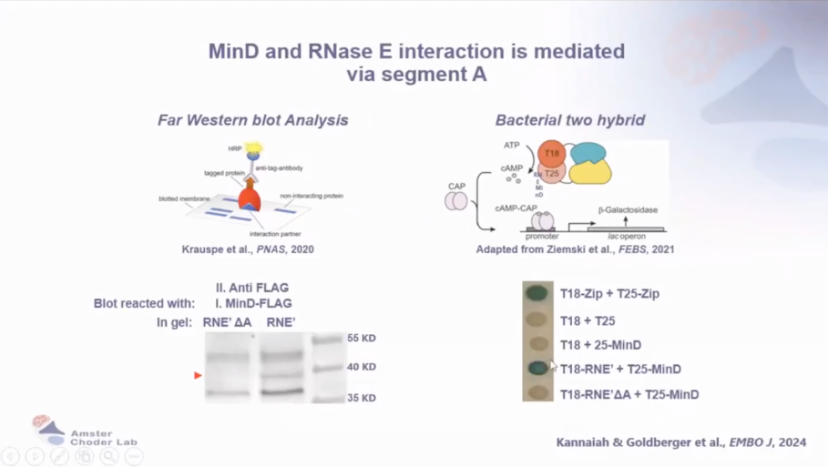
图8 MinD和RNaseE的相互作用是通过segment A介导的
3.sRNA的研究
sRNA 在感知宿主环境、促进宿主 - 微生物相互作用以及帮助细菌在宿主体内生存等方面发挥作用 。涉及多种感染类型,如肺部感染、脑膜炎、胃炎、肠道感染、皮肤感染、泌尿道感染、性传播疾病等。
sRNAs参与感知宿主环境、促进宿主-微生物相互作用和在宿主中生存等功能。
sRNAs 也能够作为信号分子,影响宿主基因的表达
sRNAs 被认为与抗生素耐药性有关。
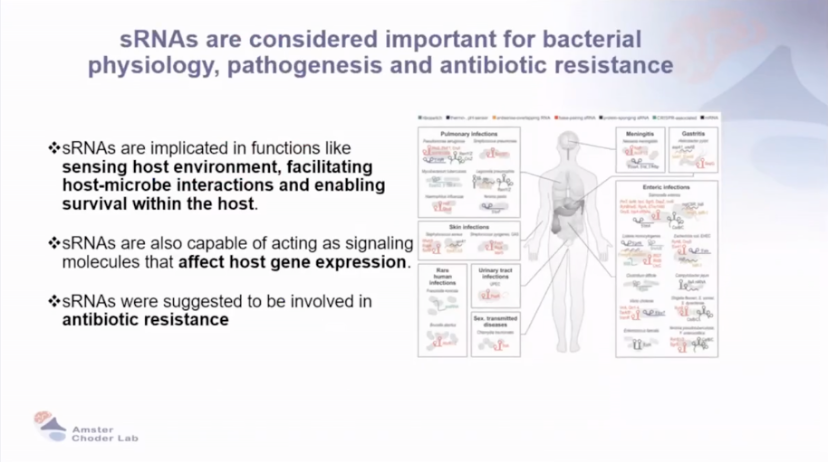
图9 sRNAs被认为对细菌的生理、致病和抗生素耐药性非常重要
三、非编码调控小RNA的组装在所有细胞类型中是一种普遍现象
1.Hfq 蛋白的功能与应激响应
Hfq作为大肠杆菌主要的 RNA 伴侣,参与 rRNA 加工、核糖体生物发生、tRNA 成熟、mRNA 翻译调控及 sRNA/mRNA 碱基配对等过程。在高渗透压、氮限制、稳定期后期等应激条件下,Hfq 蛋白在细菌两极积累。
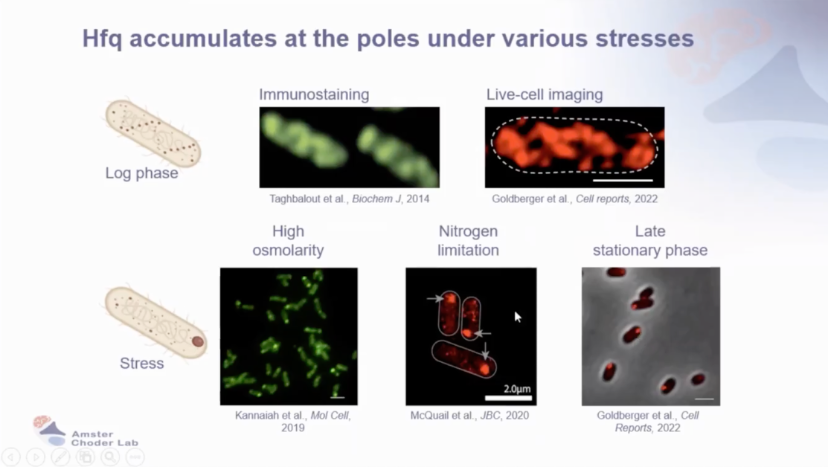
图10 Hfq在各种应力下聚集在极点
2.sRNA 介导的调控模型与相分离
应激诱导的 sRNA(核心与外周 sRNA)和 sRBP(如 Hfq)在两极形成类似 “无膜细胞器” 的结构,通过群体效应(Wisdom of the crowds)协同应对压力。图11提出 sRNA 介导的极性调控模型,探讨 Hfq 与 sRNA 的相分离可能性。
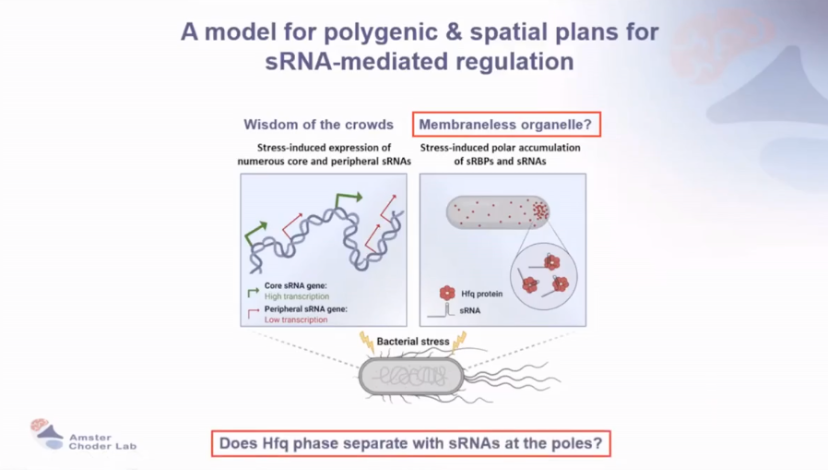
图11 sRNA介导调控的多基因与空间规划的模型
LLPS 是指分子溶液自发分离为两种不同密度的液相,形成无膜细胞器(如凝聚体),局部富集大分子并促进动态交换。核生物中,凝聚体存在于核(核仁、染色质相关凝聚体)、细胞质(应激颗粒、P 小体)和膜区(信号体)。Hfq 需与 RNA 通过异源相互作用(heterotypic interaction)发生相分离,形成含 sRNA 和 mRNA 的液滴(如 SgrS、ptsG、DsrA、rpoS 等分子共定位)。应激条件下 Hfq 形成液态凝聚体,具有动态融合 / 分裂能力,光漂白后快速恢复(FRAP 实验),应激恢复后凝聚体溶解。
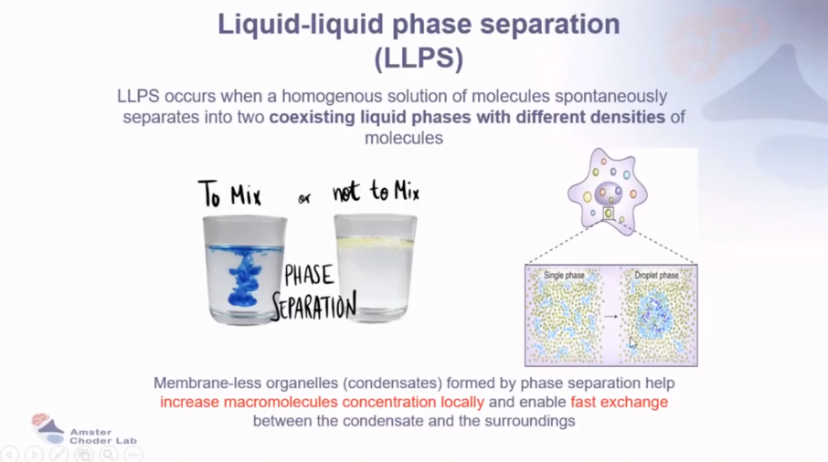
图12 液-液 相分离(LLPS)
问答环节
问题1:
极性定位蛋白的 mRNA 定位于极点,那么编码这些基因的 DNA 是否也位于极点?
回答1:
1.通过标记基因位点和 RNA,发现两者并不共定位。极点位置在细菌分裂时会变化,极性蛋白不一定靠近复制起点,因此 DNA 未必位于极点。
2.在真核生物中,RNA 可通过驱动蛋白等蛋白运输至目标位置,但细菌中可能通过非计划性机制(如降解控制)或特定蛋白(如 HFQ)实现定位,但尚未验证 HFQ 的直接作用。
3.部分蛋白(如 MreB)仅在应激条件下定位极点,而如 Ka 蛋白可能持续定位,但具体机制仍需研究。
问题2:
为何小 RNA(sRNA)在应激条件下需定位至极点?
回答2:
1.极点空间较大(染色体排除效应),可能是信号决策中心(如趋化性、糖代谢调控)。
2.蛋白(如 Morn)定位极点不依赖染色体排除,可能与膜脂成分相关(虽不依赖心磷脂,但可能有其他脂质参与)。
3.sRNA 与 HFQ 在非应激条件下也富集于极点,因拷贝数低需通过 Arlocs 技术放大检测。
问题3:
非HFQ依赖的sRNA如何定位?
回答3:
可能存在其他类似HFQ的结合蛋白辅助定位。
问题4:
革兰氏阳性菌(如链球菌)中无HFQ,sRNA如何定位?
回答4:
不同细菌可能有不同机制,需进一步研究(如链球菌的分裂平面记忆机制),未来计划合作探索。
问题5:
细菌作为更古老的生物,无膜结构分隔,极点定位是否与 RNA 结构有关?
回答5:
真核生物 RNA 定位依赖序列和结构,但细菌 sRNA 结构研究较少。其终止子结构可能参与定位,是值得探索的新方向。
问题6:
如何选择敲除的基因或 sRNA,是否具有无偏性?
回答6:
1.基于 sRNA 在极点的富集程度筛选(如选择 HFQ 结合且富集度 > 100 倍的 5 种 sRNA,如 OxyS、OmrB)。
2.选择标准为 “高频结合且高富集”,虽非随机,但旨在研究主要群体的功能,后续可扩展至其他类型 sRNA。
总结
这场报告重新审视细菌转录组织传统理论。内容围绕细菌 RNA 定位、转录与翻译关系、极性相关 RNA 定位机制、sRNA 定位及作用、RNA 伴侣蛋白研究展开,指出细菌mRNA存在细胞质、膜、极区三种定位模式,且定位独立于翻译,转录与翻译机器空间分离。还探讨了 sRNA 功能、Hfq 蛋白应激响应及相分离现象, 为未来的相关研究提供新视角。












