会议回顾
2023年12月7日,广州实验室苗智超研究员邀请了来自哈佛大学的Elena Rivas 研究员进行了题为 “ RNA结构的进化保守性(Evolutionary conservation of RNA structure)”的线上学术报告。 Elena Rivas是哈佛大学分子与细胞生物学系的高级研究员。她的实验室研究的主要重点之一是开发 RNA 结构预测的计算方法,包括识别新的 RNA 功能,开发计算筛选方法,用于识别不同生物体中进化保守的 RNA 结构。为了提高RNA功能的识别,我们至少遵循三个方向:(1)找到更好的RNA结构预测模型,以便我们能够整合结构上的先验信息,(2)开发更好的进化模型,以便我们可以有效地使用多个序列,(3) 通过评估 RNA 比对中的显着共变对来研究系统发育保守结构的检测。
本次会议中Elena Rivas 研究员报告聚焦于如何识别基因组中保守结构的概念。保守结构原则上应该具有很高的功能性和生物学相关性的可能性。我们都知道,有很多功能性的RNA,它们都具有稳定的结构。其中一个例子是RNaseP RNA,这是一种核酶(图1)。 在左边可以看到 3D结构,在右侧,可以看到它对应的二级结构(形成3D结构的碱基对)。在这里展示的特定细菌物种的结构是保守的,这种结构的保守是在细菌中和真核生物中都是保守的。虽然有小的突变,但是在序列和结构上都很容易找到这个结构领域的同源物。
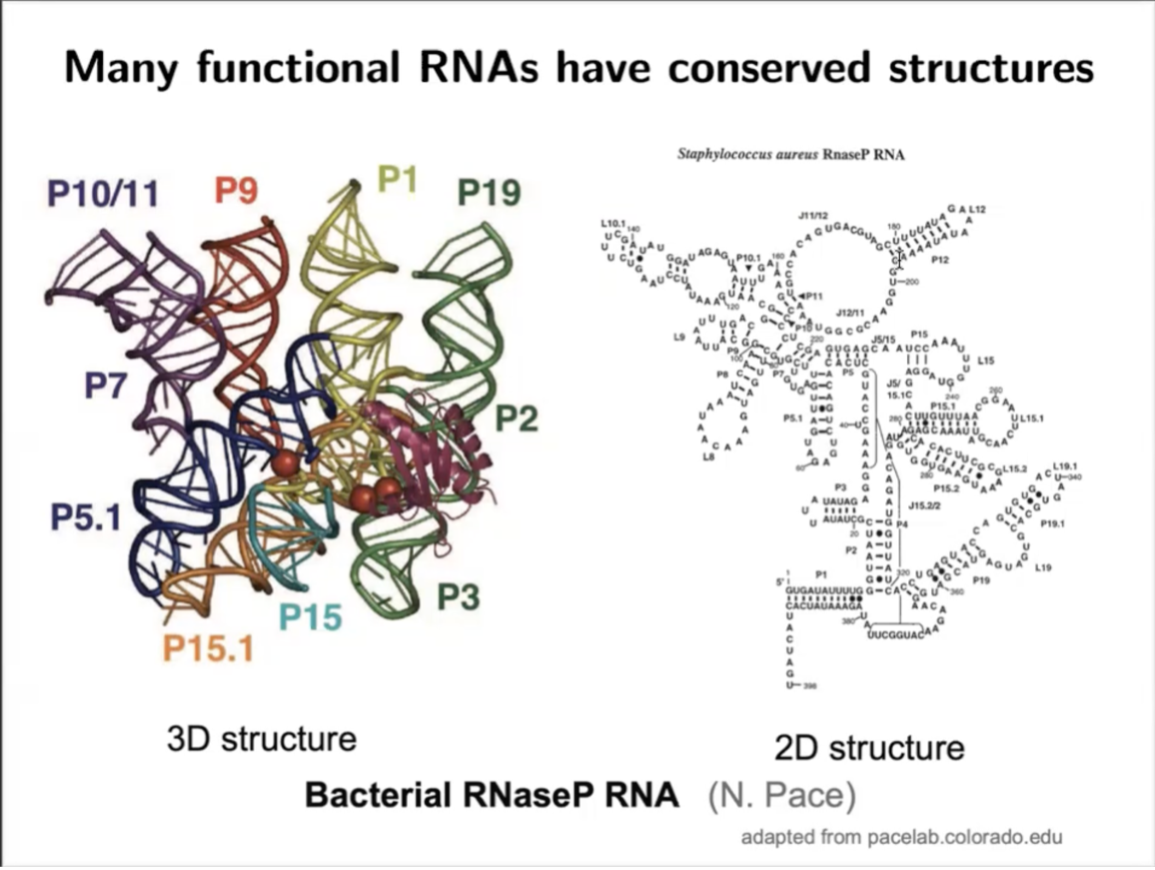
图1. RNaseP RNA
Elena Rivas 研究员提出RNA保守结构在比对中引起我们重视的是共进化。绿色的碱基标识了9个RNaseP RNA中的共进化碱基(图2)。

图2. RNA保守结构中的共进化
那么如何识别RNA结构中的共进化呢?由于R2R的局限性,这给了Elena Rivas 研究员开发一个更有力的工具的动力(图3)。RNA 碱基配对的保守性会导致序列比对中的成对共变。Elena Rivas 研究员开发了一种计算方法 R-scape(高于系统发育期望的 RNA 结构共变)可以定量测试共变分析是否支持保守 RNA 二级结构的存在(图4)。
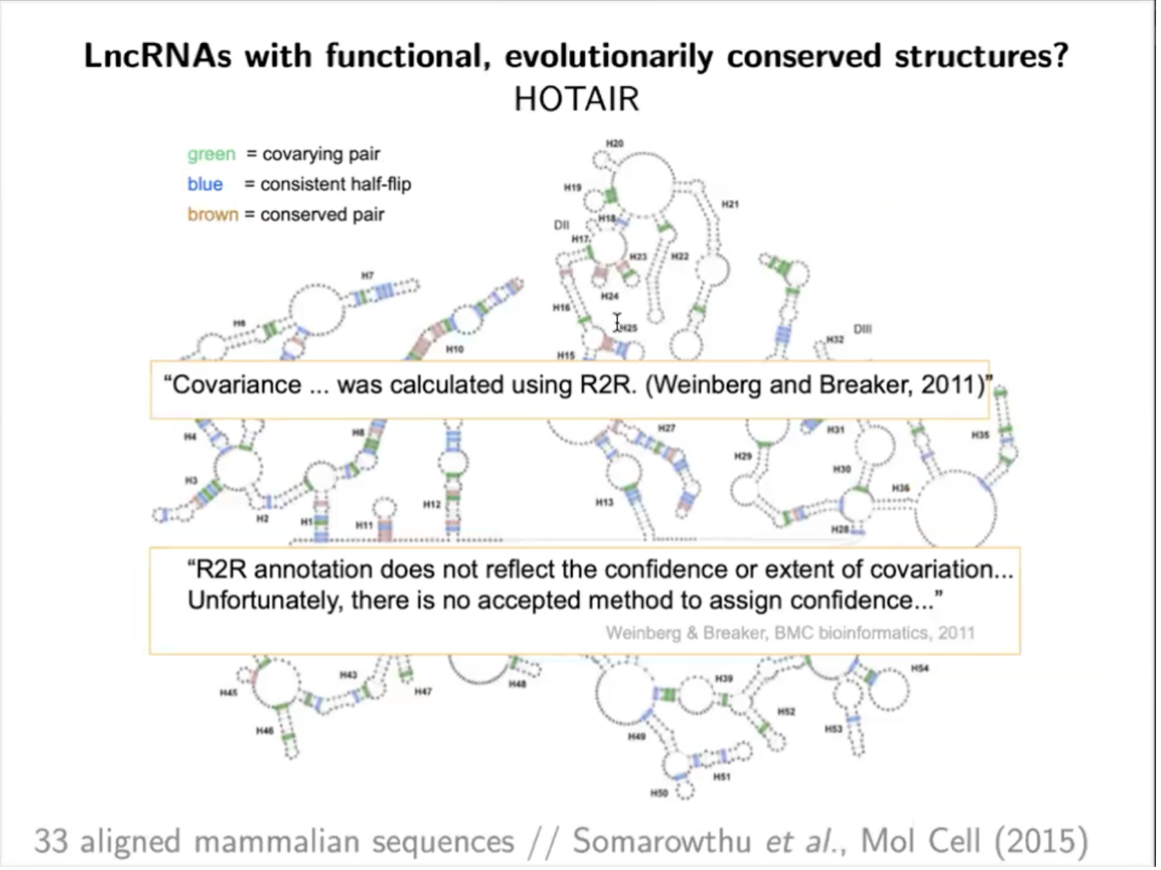
图3. R2R 的局限性

图4. Rscape RNA二级结构的共进化证据
R-scape 成功发现了传统碱基配对之外的RNA结构的元件。对比SAM-I Riboswitch 在Rfam中R2R的结果和R-scape的结果,R-scape 成功发现了假结结构(图5)。
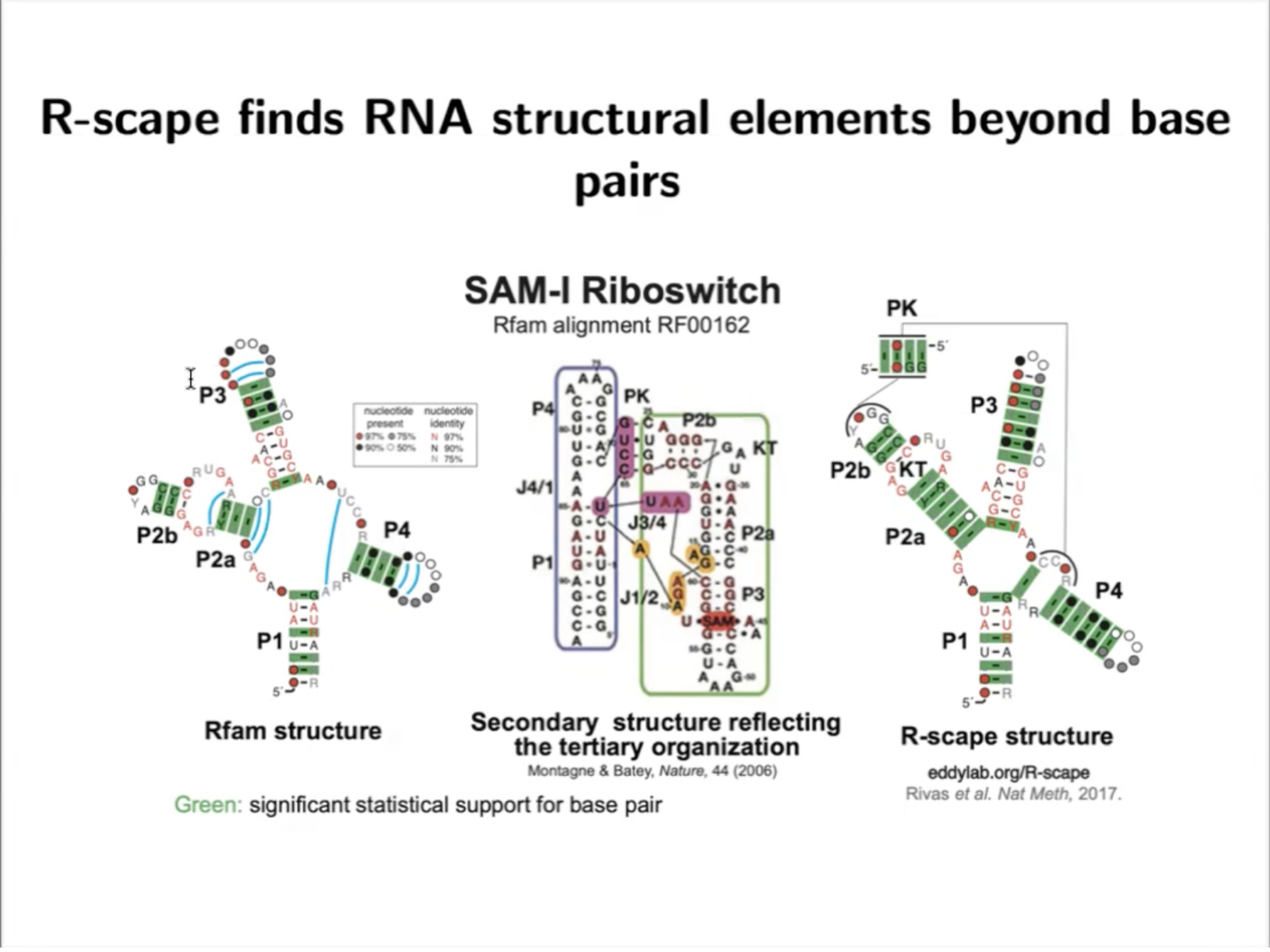
图5. R-scape 成功发现SAM-I核糖开关中的假结结构
在此之后,基于共进化的信息,Elena Rivas 研究员开发了RNA结构预测软件Cacofold。 因为好的共进化信息可告诉我们哪些配对是应该包含的,哪些是不该包含的。
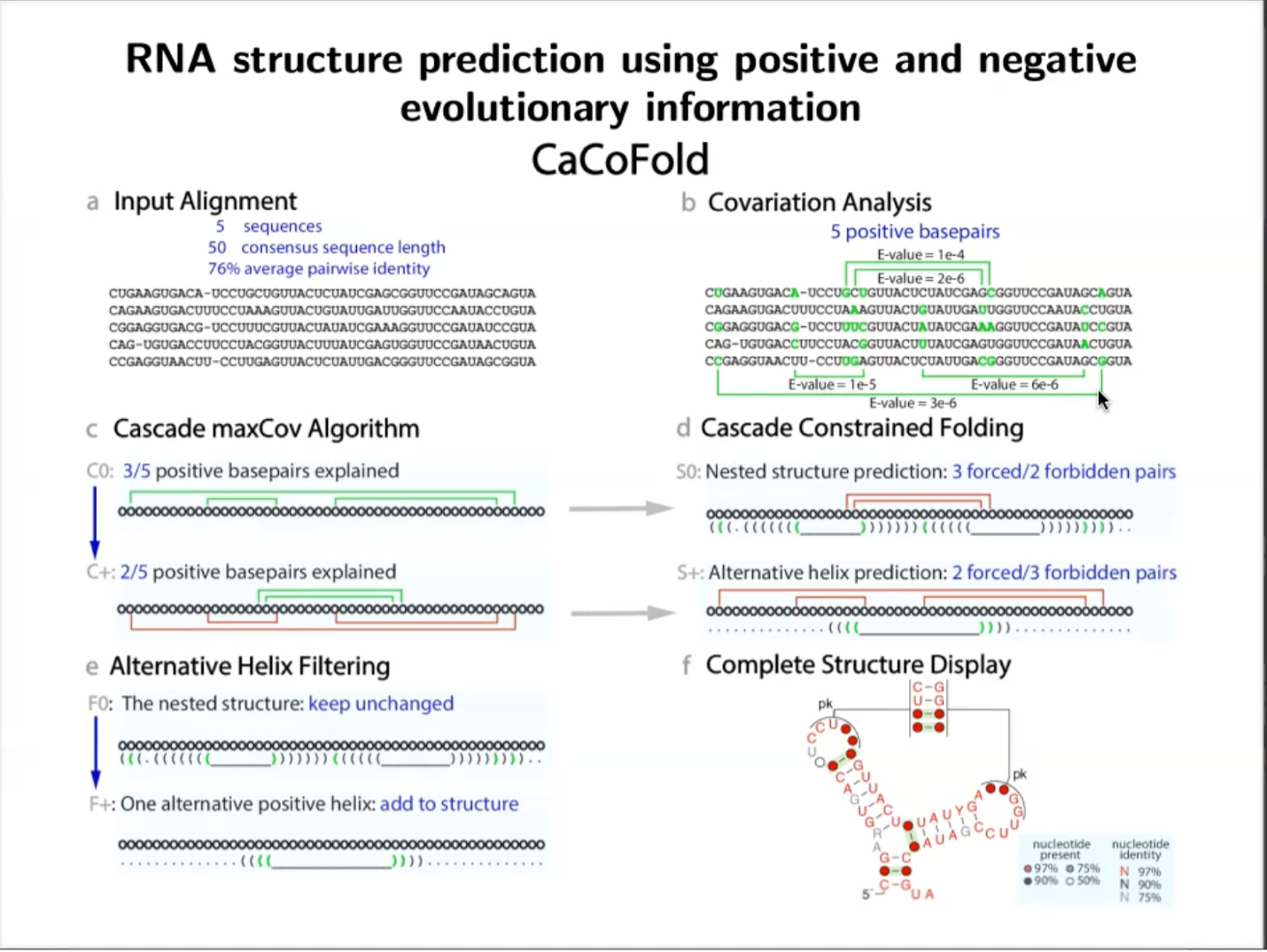
图6. CaCoFold 流程图
总结,Elena Rivas 研究员告诉我们的就是RNA结构中的共进化信息可以带给我们很多结构上的信息。Elena Rivas 研究员今天使用的所有的信息仅仅是序列的对齐,通过序列的对齐来发现共进化的信息,进而发现RNA结构中新的碱基配对,甚至预测更准确的结构,没有其他的输入。
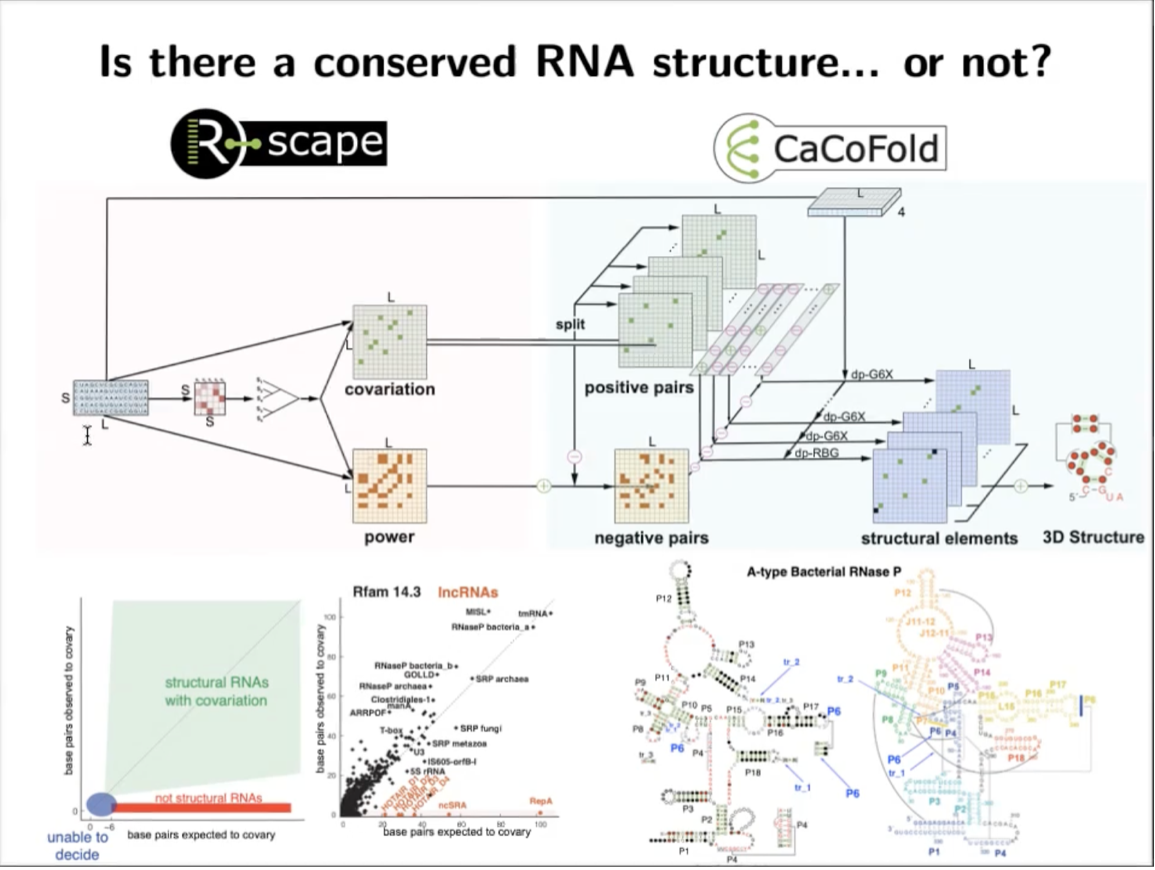
图7. RNA序列中的共进化信息能带给我们什么
之后进行了讨论,总结关键的问题如下:
问题1:rMSA,是由 Anna Marie Pyle开发的多序列比对程序。我想你解释了为什么它表现的不够好,但大多数程序都会使用它,因为它是自动化的并且它自己声称它表现的很好。 因此,当我们没有更多选择时,你认为我们可以使用它吗?
Elena Rivas 研究员回答道:你可以使用hammer。如果你正在寻找一个结构,你就不能推断一个结构。你不能提出一个结构并符合该结构。这就是统计学中所谓的双DP。举个例子,如果你对HOTAIR做对齐,你不会得到任何信息,因为没有一致的共进化信息,但是如果你对COOLAIR做对齐,你可以做到这一点,它到处都是一种变化,COOLAIR是一个非常保守的序列,因为它对蛋白质协调的作用很重要,所以对齐真的很深。
问题2:lncRNA 没有好的三级结构是么?
答:我们现在没有观察到很多多样性,所以我们很难找到共进化的信息来确定保守的结构,我们可能需要更好的工具。
Elena Rivas 研究员此次会议报告已收录于Guangzhou RNA club bilibili视频网站(https://www.bilibili.com/video/BV1HT4y187b8/?spm_id_from=333.999.0.0)
欢迎关注Guangzhou RNA club公众号、网站(rnaclub.rnacentre.org)、twitter(@RNA_club)。






