会议回顾
2023年10月25日,广州实验室苗智超研究员邀请了Matthias W. Hentze教授进行标题为“核糖调节剂:生物学原理、作用机制与应用前景”的线上学术报告。
Matthias W. Hentze教授是欧洲分子生物学实验室(EMBL)主任,海德堡大学医学系教授。其课题组结合生化和系统级方法来研究基因表达与细胞代谢之间的联系,及它们在人类疾病中的作用,该小组特别关注RNA生物学和RNA结合蛋白。目前致力于通过RNA结合蛋白(RBP)和非编码调控RNA调控mRNA在细胞质中执行控制基因表达的重要步骤。未来目标在于探索、定义和理解enigmRBP和REM网络,并阐明RNA代谢在疾病中的作用,基于此开发新型诊断和治疗策略等。
摘要
目前通过无偏见的全系统分析,已找到了数万个RNA结合蛋白(RBP),其中之前已知的RBP还不到一半。这一令人惊讶的结果带来了一系列新的挑战,如:蛋白中非规则结构的RNA结合区具有RNA-蛋白凝聚作用;中间代谢产物和RNA功能调控之间存在普遍的潜在联系。
核糖调节(riboregulation)作为生物调控机理的新范式,通过RNA来直接调控蛋白质功能。我们开始去理解核糖调节的分子机制,通过对小鼠器官中RBP的研究,揭示了它们在哺乳动物生理学中的广泛作用。我将讨论RBP世界的这些新发现及其对细胞生物学、代谢和疾病机制的影响。
会议内容
Matthias介绍的报告内容包括核糖调控的原理、生物学机制和研究范围。谈论的许多的方面对于RNA研究人员来说甚至是相当不寻常和反教条的。
本次报告主要探讨了RNA在生物调控中的新角色——核糖调控。Matthias首先介绍了细胞调控的基本原则,即通过控制蛋白质之间的相互作用和酶与底物之间的相互作用来控制细胞活动。然后,Matthias提出了一个新观点,即RNA也可以通过类似于翻译后修饰的方式来调控蛋白质和酶底物之间的相互作用。
具体来说,Matthias介绍了RNA结合蛋白(RNA binding protein,RBP)在核糖调控中的作用。传统上,人们认为RBP是通过结合RNA来调控RNA的命运和功能的。但是,Matthias指出,RBP本身也可以被RNA分子所影响,从而导致蛋白质功能的变化。这种调控的机制类似于翻译后修饰,但它是由RNA分子来实现的。
为了支持这一观点,Matthias介绍了一些实验结果。例如,他们使用了RNA相互作用捕获技术来识别与RNA相互作用的RBP (RNA 结合蛋白),并发现这些RBP可以调控多种生物学过程。此外,Matthias还介绍了一些具体的例子。
常见的生物学调控机制
图1 展示了生物学被调节的两种最常见的方式。如图1所示,一个方式是控制蛋白质相互作用,另一个方式是控制酶和它们的底物之间的相互作用。几十年来的研究通过翻译后修饰的方式确定了蛋白质调节过程。而RNA可以做与翻译后修饰在控制蛋白质和酶底物相互作用方面完全相同或类似的事情。即使在2023年,这也是一个相当不寻常的想法。对于一个经典的RNA结合蛋白,大多数人的看法是,这些蛋白通常含有RND结合结构域,如RM结构域,KH结构域等几个特征良好的结构域之一。这些蛋白与RNA、信使RNA、非编码RNA等结合,并改变该RNA命运等。我们会认为,剪接调节器控制剪接过程,翻译调节器控制翻译过程。它们将RNA带到树突的特定空间位置。换言之,蛋白质控制着它结合的RNA。
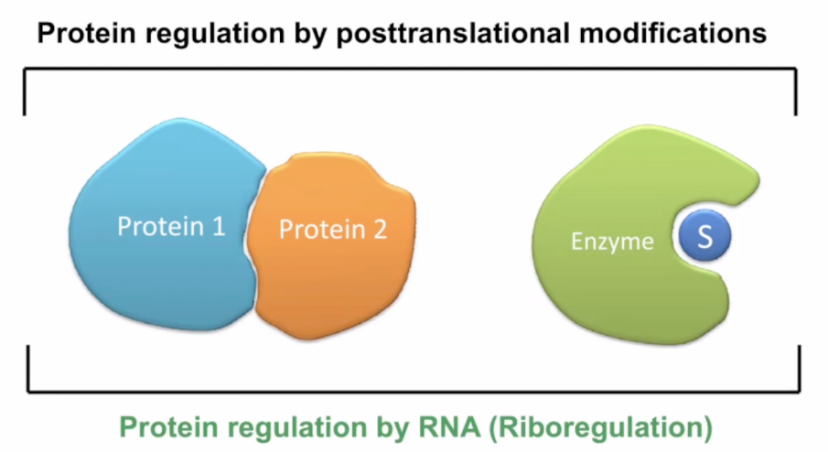
图1. 最常见的两种生物学调控方式
核糖调控机制
与常见的蛋白质主导的生物调控机制相反,在核糖调节机制中,RNA控制蛋白质的命运并改变蛋白质功能。这种类型的RNA结合蛋白通常是没有RNA结合域的蛋白质。实际上,它们甚至通常不被称为RNA结合蛋白。它们更有可能被称为酶或先天免疫效应分子作为通道或溶质载体等其他东西。因此,这与我们传统上对RNA结合蛋白及其功能的看法非常不同。
Matthias还与大家分享他对"moonlighting" (兼职)这个术语的理解。生物学中的兼职指的是蛋白质或基因在细胞或有机体中具有多种功能或作用的现象。这意味着一个特定的蛋白质或基因可以在不同的细胞或生理环境中执行不同的任务或具有不同的功能。这一概念突出了生物系统的多功能性和复杂性。
"moonlighting"与"Riboregulation"是不同的概念。"moonlighting"和"Riboregulation"的共同之处在于RNA结合蛋白通常具有其他功能,例如作为酶。但是在"moonlighting"中,RNA结合蛋白(可以是酶)像传统的RNA结合蛋白一样结合到RNA上,并改变其命运。在我最早研究的RNA生物学中,RNA结合蛋白铁调节蛋白1(IRP1)就是一个例子,它是一种酶,同时又作为一个RNA结合蛋白调控铁蛋白的翻译或透明受体信使RNA的稳定性,这是现在的教科书知识。
然而,兼职与核糖调控不同(见图2)。Matthias提出了一个重要的观点,riboregulation。虽然这对Matthias的团队来说是成为一个核心话题,Matthias的团队也是作为这一普遍原则推广的人。但早期就有了一些与riboregulation类似的例子。所以如果我们想到PKR(蛋白激酶R),就不应该感到太惊讶了。PKR是一种在双链RNA存在下被激活的蛋白激酶,在病毒感染的情况下通常会被激活。然后PKR在激活后磷酸化翻译起始因子α,这会下调整体翻译作为先天免疫反应的重要方面。一些类似受体也能够感知RNA并被单链或双链RNA激活的分子。
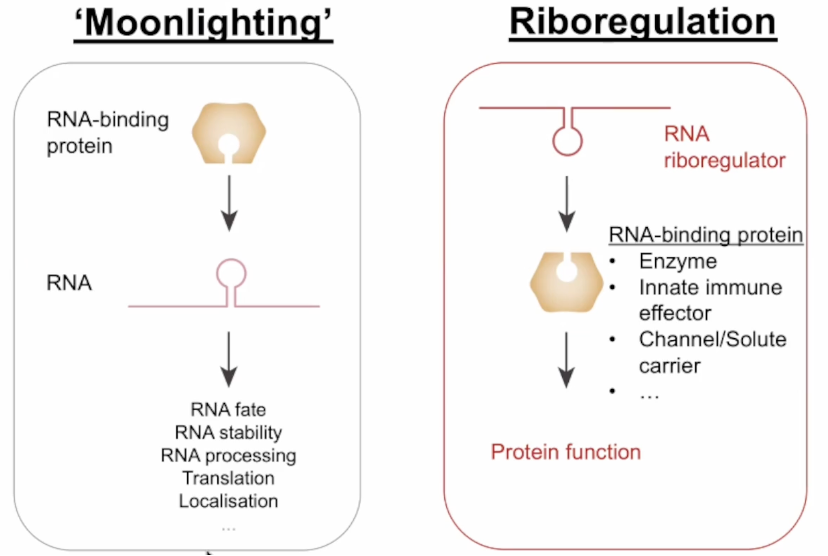
图2. 两种相互作用机制:moonlighting 与 riboregulation
系统性地发现新的RNA结合蛋白
在细菌中,CSRNA在2000年被认为可以控制大肠杆菌RNA聚合酶的活性。即使过去有例子,但一般原则没有被认可。但这是一个说明riboregulation 机制的重要的实体。十多年前,Matthias的实验室开发了一种叫做RNA相互作用捕获的流程,可以系统性地获的大量的RNA结合蛋白。现在,这个流程不断被优化,甚至可以在活细胞研究中全面发现所有活性RNA结合蛋白。
这个实验的原理是,用波长为254纳米的紫外光来照射组织切片,让RNA和直接结合该RNA的蛋白质之间形成共价键。图3所示的椭圆,代表蛋白,红色椭圆表示的蛋白在紫外线照射时,与所示的RNA共价交联。值得注意的是,这种类型的紫外线照射不会诱导蛋白质-蛋白质交联,因此图中灰色椭圆不会与红色椭圆交联,这意味着我们可以特异性地捕获与RNA直接结合的蛋白。
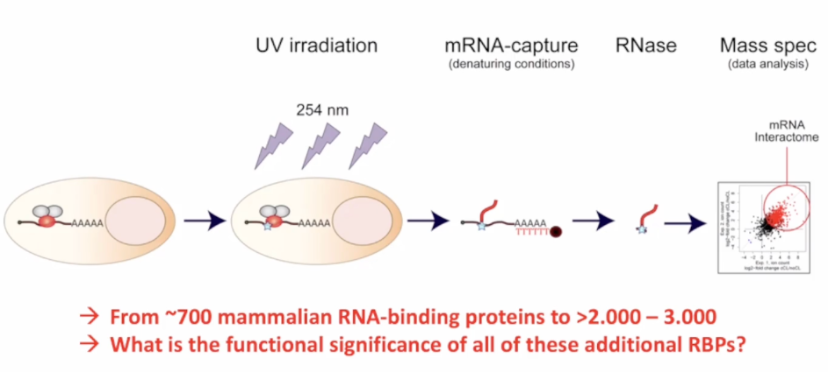
图3. 系统性地发现RNA结合蛋白
然后,可以使用寡聚dT寡核苷酸来检出带有poly-A尾巴的信使RNA(如信使RNA或一些众所周知的长编码RNA),并通过洗涤剂、变性剂和高盐洗涤等方法去除与RNA没有共价结合的物质,基本上只保留交联的RNA结合蛋白质(RBPs),然后通过质谱法释放并鉴定它们。当进行这项工作时,Matthias的团队很快将已知的RNA结合蛋白质数量从以前的大约七八百个增加到了一个意料之外的数量范围内。发现了2000多个RNA结合蛋白,并逼近3000个。
这个发现引发了许多问题。他们认识到的许多额外蛋白质并没有RNA结合结构域,所以这些蛋白质为什么会与RNA结合是无法解释的。同时也没有与RNA生物学相关的已注释生物学功能,所以也许这些蛋白与RNA无关,只是偶然发生的现象。然而,这些蛋白质并不像人们最初怀疑的那样只是常见的污染物。实际上,将这些RNA结合蛋白质与全蛋白组学分析进行比较后,Matthias的团队发现它们在各种丰度水平上都存在,包括非常稀有的蛋白质,并且许多特异性控制实验证明,在生物体内至少在生化水平上存在特异性的RNA结合蛋白质。
这是很有趣的现象,但对于生物学家来说更重要的问题是它是否具有功能意义。因此,Matthias的实验室主要工作重点是解决这个问题。这些蛋白质与RNA结合的功能意义是什么?Matthias介绍了两个已经发表的例子。
p62与VtRNA 1-1 之间相互作用调控自噬通路
首先讲述的是,Matthias团队中的Rusty Horrors在实验室中研究的一个例子。Rusty的项目挑战是:在新发现的长长的RNA结合蛋白清单中,是否有一个已经有很多发表研究的蛋白质,并且已发表的论文中,不应该有关于RNA结合或RNA生物学功能的证据,因为这应该是一个在实验中稳健地显示为RNA结合蛋白质的蛋白质。实际上,Matthias要求Rusty不要把最好的蛋白放在前面,而是把最差的蛋白放在前面,即以前从未被发现与RNA有任何关系的蛋白。
于是他选择了p62蛋白。这是哺乳动物自噬通路中涉及的一种蛋白质,在当时已经发表了超过4000篇论文,其中没有一篇论文提到RNA的重要性,正如这篇论文所报道的那样。Rusty发现这种蛋白质并没有RNA结合域,但可以结合一个小的非编码RNA(如图4所示),称为vtRNA1-1。这是一种RNA聚合酶转录本,类似tRNA分子,人类基因组中有四种不同的vtRNA,只有其中一种在自噬途径中起作用。
由于寡聚化对于促进自噬至关重要,正如图4所示,vtRNA 1-1是一个抑制剂,通过抑制蛋白质之间的相互作用来抑制自噬。而且,重要的是,vtRNA 1-1的水平本身受到饥饿的调控。通常,饥饿是触发自噬的信号,当细胞饥饿时,vtRNA 1-1的水平会下调。与此RNA结合的p62单体的数量显然会下降。单体被释放出来后可以聚合并促进自噬通量。因此,这个例子突出了一个riboregulation调控的例子,其中一个非常特定的RNA(vtRNA 1-1)控制着蛋白质相互作用,通过调节RNA的稳态水平来实现生物调控。这个例子提出了三个后续问题:
1. 为什么vtRNA如此特殊?为什么这个特定的RNA能够起作用,而其他vtRNA则不能?
2. P62没有RNA结合结构域,这个RNA是如何结合的?
3. RNA和蛋白质之间的相互作用如何抑制聚合?
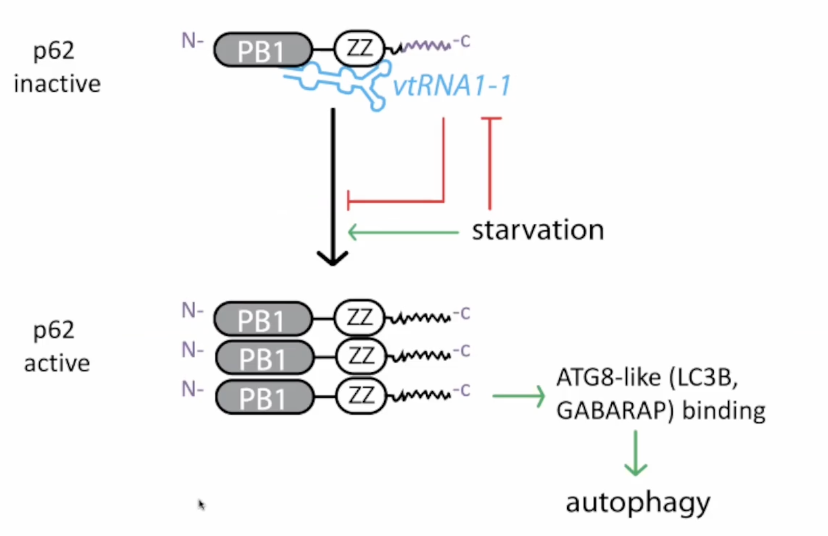
图4. 非编码RNA调控细胞自噬
vtRNA 1-1 重要的特点是它具有中央柔性环,而其他三个vtRNA没有。当在这个环中进行突变时,vtRNA失去了对p62的结合能力,并且该突变体失去了调节能力。因此,这显然是RNA特异性结合的一个重要方面。P62结合到RNA的末端端部,即所谓的PB1结构域。如图5右侧所示,PB1结构域在自噬领域的研究人员中已经非常出名,因为它是一个重要的结构域。其正电荷的K7和R21与另一个亚单位的负电荷的所谓OPCA基序结合,通过电荷驱动相互作用进行聚合。正电荷区域K7和R21也是关键的结合位点,这是通过vtRNA 1-1的突变分析确定的。
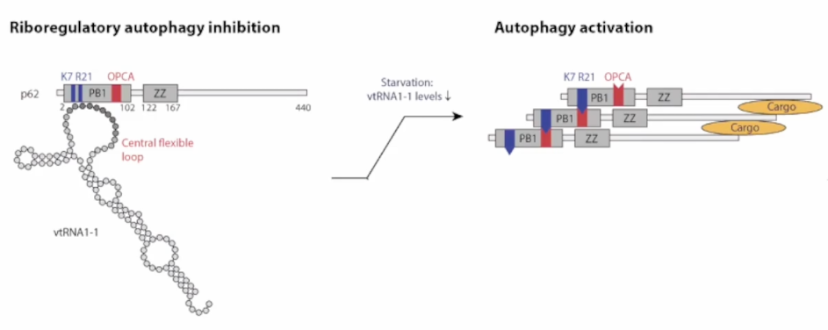
图5. p62与VtRNA 1-1 通过相互作用调控自噬通路
受到乙酰化控制的核糖调节
接着,Matthias介绍了另外一个非常不同的例子,旨在展示核糖调控发生机制的多样性。这个例子是人类糖酵解酶Enolase 1。相信大多数人在生物化学课程中都很熟悉Enolase,因为它催化了糖酵解的倒数第二步。Ina Hoopers曾经是一名博士后,他发现Enolase的酶活性也受到核糖调控。在图6中,可以看到从数据库中下载的Nlas的晶体结构(其他团队上传的结构)。可以看到它作为一种酶的活性形式,并且可以看到Matthias突出显示了四个赖氨酸残基,之所以突出显示它们是因为发现这些残基的乙酰化可以以一种生物调控的方式激活NLS的RNA结合活性。
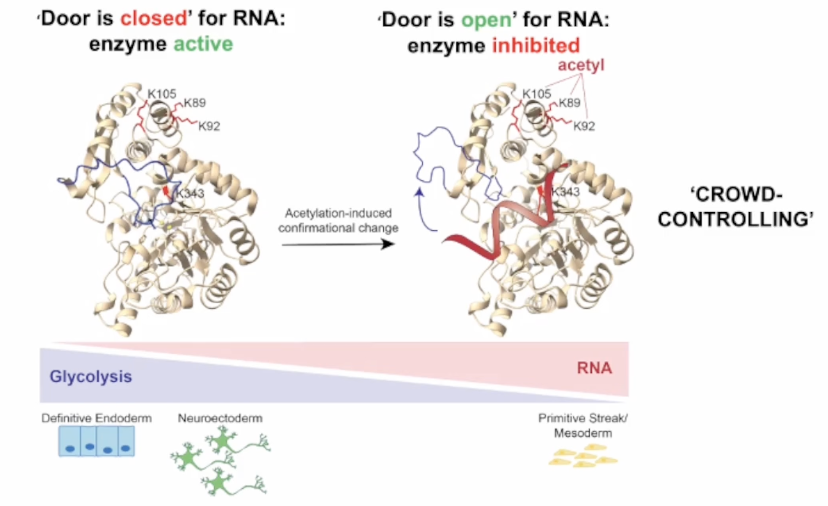
图6. 受到乙酰化控制的核糖调节
这只是一个工作模型,暂时还没有结构数据。设想一下,例如在活性酶中覆盖酶中心的蓝色环可能会摆动开放,但也可能是其他任何激活RNA结合的方式。但乙酰化会激活RNA结合,并且激活RNA结合发生在靠近酶催化中心的位置,如图6中红色标注的K343所示。突出显示它是因为如果将这个赖氨酸突变为天冬氨酸并制备重组突变蛋白,则这种突变分析与野生型蛋白一样活跃。因此,它本身不会影响酶活性。但是当测试RNA结合活性时,这种突变分析显示出强烈减少的RNA结合能力。因此,它是研究RNA结合重要性的良好突变模型。当向这种突变的酶中添加 RNA时,RNA并不起作用,但当我们向野生型酶中添加RNA时,则会抑制Enolase的酶活性。这在体外简单的还原生化实验中和活细胞中都会发生。
关于结合的RNA,与先前展示的p62的情况不同,尽管此处的结合也是特异性的。在这种情况下,约有2000个不同的片段主要包含在细胞转录因子的非编码区域中,Matthias的团队已经合成了这些区域的35个核苷酸的短片段。当他们只使用这些合成的35个核苷酸片段时,在体外观察到酶抑制作用,而当把这些35个核苷酸片段引入活细胞中时,这些特定的35个核苷酸片段会下调这些活细胞中的糖酵解作用。这个相互作用是特异性的,因为可以制作这些35个核苷酸片段的乱序版本时,完全没有起作用。
随后,Matthias将这些突变引入小鼠胚胎干细胞的基因组中,以便它们表达具有改变RNA结合能力的突变Enolase或野生型Enolase。大多数胚胎干细胞可以分化为不同的生殖层,而突变体无法正确分化,并且无法下调糖酵解作用(糖酵解是分化中胚胎干细胞代谢的一个重要方面)。
总的来说,本次报告提出了一个新颖的观点,即RNA可以通过竞争性调控机制来影响蛋白质和酶底物之间的相互作用。这一观点挑战了传统的思维方式,并为RNA研究者提供了一个全新的思考方向。Matthias通过实验结果和具体例子来支持这一观点,并强调了RNA在生物调控中的重要性。
参考文献
1. Castello, A., B. Fischer, K. Schuschke, R. Horos, B.M. Beckmann, C. Strein, N.E. Davey, D.T. Humphreys, T. Preiss, L.M. Steinmetz, J. Krijgsveld and M.W. Hentze. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell 149, 1393-1406, 2012.
2. Beckmann, B.M., R. Horos, B. Fischer, A. Castello, K. Eichelbaum, A.-M. Alleaume, T. Schwarzl, T. Curk, S. Foehr, W. Huber, J. Krijgsveld and M.W. Hentze. The RNA-binding proteomes from yeast to man harbor conserved enigmRBPs. Nature Commun. 6:10127, DOI:10.1038/ncomms10127, 2015.
3. Castello, A., B. Fischer, C.K. Frese, R. Horos, A.-M. Alleaume, S. Foehr, T. Curk, J. Krijgsveld and M.W. Hentze. Comprehensive identification of RNA-binding domains in human cells. Mol. Cell 63, 696-710, 2016.
4. Hentze, M.W., A. Castello, T. Schwarzl and T. Preiss, A brave new world of RNA-binding proteins. Nature Rev. Mol. Cell Biol. 19, 327-341, 2018.
5. Horos, R., M. Büscher, R. Kleinendorst, A.-M. Alleaume, A.K. Tarafder, T. Schwarzl, D. Dziuba, C. Tischer, E.M. Zielonka, A. Adak, A. Castello, W. Huber, C. Sachse and M.W. Hentze, The small non-coding vault RNA1-1 acts as a riboregulator of autophagy. Cell 176, 1054-1067, 2019.
6. Huppertz, I., J.I. Perez-Perri, P. Mantas, T. Sekaran, T. Schwarzl, F. Russo, D. Ferring-Appel, L. Dimitrova-Paternoga, E. Kafkia, J. Hennig, P.A. Neveu, K. Patil and M.W. Hentze. Riboregulation of Enolase 1 Activity Controls Glycolysis and Embryonic Stem Cell Differentiation. Mol. Cell 82, 2666-2680, 2022.
7. Perez-Perri, J.I., D. Ferring-Appel, I. Huppertz, T. Schwarzl, F. Stein, M. Rettel, B. Galy and M.W. Hentze. The RNA-binding protein landscapes differ between mammalian organs and cultured cells. Nature Comms. 14:2074, https://doi.org/10.1038/s41467-023-37494-w, 2023.
Matthias W. Hentze教授此次会议报告已收录于Guangzhou RNA club bilibili视频网站(https://www.bilibili.com/video/BV1Hw411q7cf/?spm_id_from=333.999.0.0)
欢迎关注Guangzhou RNA club公众号、网站(rnaclub.rnacentre.org)、twitter(@RNA_club)。





